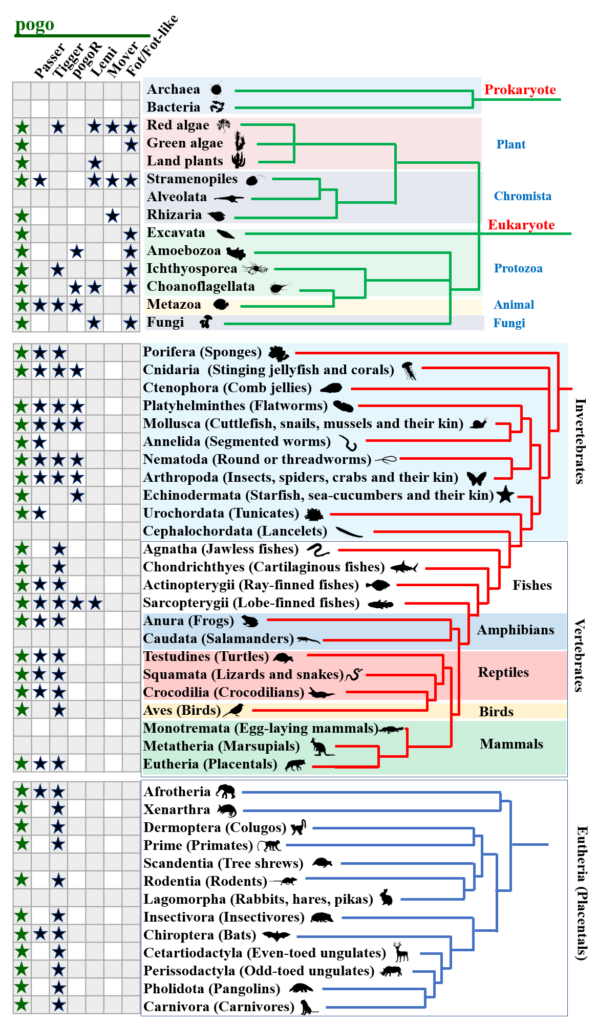
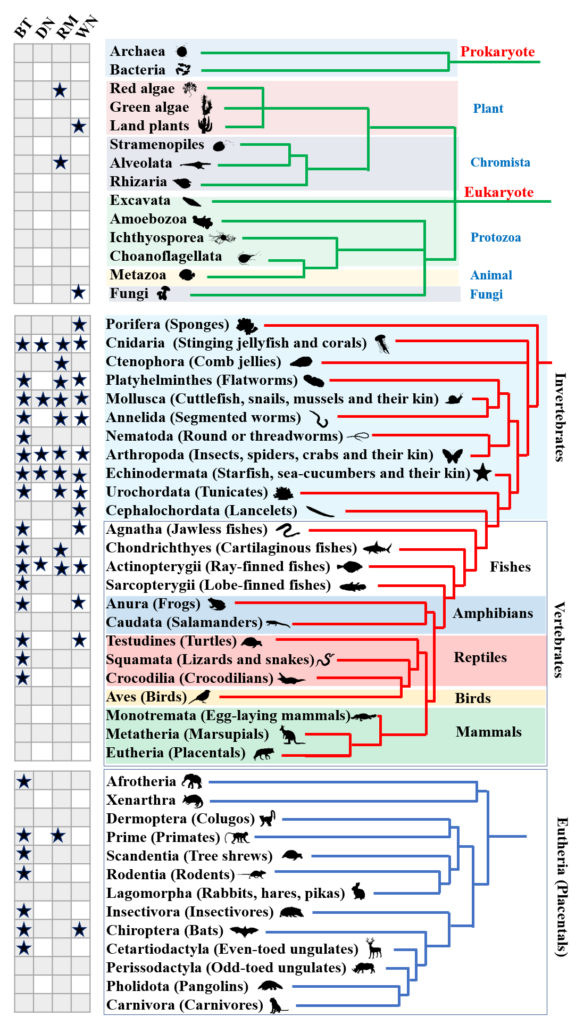
Transposable elements, contributors in the evolution of organisms (from an arms race to a source of raw materials), Heliyon. 2021. PMID: 33532648.
Specificities and Dynamics of Transposable Elements in Land Plants. Biology (Basel). 2022. PMID: 35453688
A Field Guide to Eukaryotic Transposable Elements. Annu Rev Genet. 2020. PMID: 32955944.
A survey of transposable element classification systems–a call for a fundamental update to meet the challenge of their diversity and complexity. Mol Phylogenet Evol. 2015. PMID: 25797922.
All Quiet on the TE Front? The Role of Chromatin in Transposable Element Silencing. Cells. 2022. PMID: 36010577.
An epigenetic toolkit allows for diverse genome architectures in eukaryotes. Curr Opin Genet Dev. 2015. PMID: 26649755.
Bridging multiple dimensions: roles of transposable elements in higher-order genome regulation. Curr Opin Genet Dev. 2023. PMID: 37028152.
Coevolution between transposable elements and recombination. Philos Trans R Soc Lond B Biol Sci. 2017. PMID: 29109221.
Constitutive Heterochromatin in Eukaryotic Genomes: A Mine of Transposable Elements. Cells. 2022. PMID: 35269383.
Dark genome, bright ideas: Recent approaches to harness transposable elements in immunotherapies. Cancer Cell. 2022. PMID: 35907399.
Distinguishing friends, foes, and freeloaders in giant genomes. Curr Opin Genet Dev. 2018. PMID: 29544119.
Diversity and evolution of transposable elements in Arabidopsis. Chromosome Res. 2014. PMID: 4801342.
Double-edged sword: The evolutionary consequences of the epigenetic silencing of transposable elements. PLoS Genet. 2020. PMID: 32673310.
Epigenetic regulation of intragenic transposable elements: a two-edged sword. J Biochem. 2018. PMID: 30010918.
Evolution of transposable elements and evolution of eukaryote genomes mediated by transposable elements. Genes Genet Syst. 2019. PMID: 31996493.
Evolutionary impact of transposable elements on genomic diversity and lineage-specific innovation in vertebrates. Chromosome Res. 2015. PMID: 26395902.
Giant Transposons in Eukaryotes: Is Bigger Better? Genome Biol Evol. 2019. PMID: 30796812.
Horizontal acquisition of transposable elements and viral sequences: patterns and consequences. Curr Opin Genet Dev. 2018. PMID: 29505963.
Horizontal transfers of transposable elements in eukaryotes: The flying genes. C R Biol. 2016. PMID: 27234293.
Host Gene Regulation by Transposable Elements: The New, the Old and the Ugly. Viruses. 2020. PMID: 32993145.
Host-transposon interactions: conflict, cooperation, and cooption. Genes Dev. 2019. PMID: 31481535.
How retrotransposons shape genome regulation. Curr Opin Genet Dev. 2016.PMID: 26855260.
Impact of transposable elements on the evolution of complex living systems and their epigenetic control. Biosystems. 2021. PMID: 34718084.
Integration of TE Induces Cancer Specific Alternative Splicing Events. Int J Mol Sci. 2022. PMID: 36142830.
Integration site selection by retroviruses and transposable elements in eukaryotes. Nat Rev Genet. 2017. PMID: 28286338.
Investigating the Potential Roles of SINEs in the Human Genome. Annu Rev Genomics Hum Genet. 2021. PMID: 33792357.
Jumping the fine LINE between species: horizontal transfer of transposable elements in animals catalyses genome evolution. Bioessays. 2013. PMID: 24003001.
Mammalian genome evolution as a result of epigenetic regulation of transposable elements. Biomol Concepts. 2014. PMID: 25372752.
Mammalian genome innovation through transposon domestication. Nat Cell Biol. 2022. PMID: 36008480.
Mammalian transposable elements and their impacts on genome evolution. Chromosome Res. 2018. PMID: 29392473.
Massive contribution of transposable elements to mammalian regulatory sequences. Semin Cell Dev Biol. 2016. PMID: 27174439.
Methodologies for the De novo Discovery of Transposable Element Families. Genes (Basel). 2022. PMID: 35456515.
Mobility connects: transposable elements wire new transcriptional networks by transferring transcription factor binding motifs. Biochem Soc Trans. 2020. PMID: 32573687.
More than causing (epi)genomic instability: emerging physiological implications of transposable element modulation. J Biomed Sci. 2021. PMID: 34364371.
New Insights on the Evolution of Genome Content: Population Dynamics of Transposable Elements in Flies and Humans. Methods Mol Biol. 2019. PMID: 31278675.
On the Importance to Acknowledge Transposable Elements in Epigenomic Analyses. Genes (Basel). 2019. PMID: 30935103
Recent Bioinformatic Progress to Identify Epigenetic Changes Associated to Transposable Elements. Front Genet. 2022. PMID: 35646069.
Regulatory activities of transposable elements: from conflicts to benefits. Nat Rev Genet. 2017. PMID: 27867194
Repetitive elements in aging and neurodegeneration. Trends Genet. 2023. PMID: 36935218.
Retrotransposons as a Source of DNA Damage in Neurodegeneration. Front Aging Neurosci. 2022. PMID: 35058771.
Rewiring of chromatin state and gene expression by transposable elements. Dev Growth Differ. 2021. PMID: 34050925.
Role of Transposable Elements in Gene Regulation in the Human Genome. Life (Basel). 2021. PMID: 33557056.
Role of Transposable Elements in Genome Stability: Implications for Health and Disease. Int J Mol Sci. 2022. PMID: 35887150.
Roles of Transposable Elements in the Different Layers of Gene Expression Regulation. Int J Mol Sci. 2019. PMID: 31731828.
Roles of transposable elements in the regulation of mammalian transcription. Nat Rev Mol Cell Biol. 2022. PMID: 35228718.
Silencing of Transposable Elements by piRNAs in Drosophila: An Evolutionary Perspective. Genomics Proteomics Bioinformatics. 2017. PMID:28602845.
Structural and sequence diversity of eukaryotic transposable elements. Genes Genet Syst. 2020. PMID: 30416149.
Taming the Turmoil Within: New Insights on the Containment of Transposable Elements. Trends Genet. 2020. PMID: 32473745.
Taming transposable elements in vertebrates: from epigenetic silencing to domestication. Trends Genet. 2022. PMID: 35307201.
Taming, Domestication and Exaptation: Trajectories of Transposable Elements in Genomes. Cells. 2021. PMID: 34944100.
TFs for TEs: the transcription factor repertoire of mammalian transposable elements. Genes Dev. 2021. PMID: 33397727.
The challenges of predicting transposable element activity in hybrids. Curr Genet. 2021. PMID: 33738571.
The evolution and function of transposons in epigenetic regulation in response to the environment. Curr Opin Plant Biol. 2022. PMID: 35961279.
The Evolutionary Volte-Face of Transposable Elements: From Harmful Jumping Genes to Major Drivers of Genetic Innovation. Cells. 2021. PMID: 34831175.
The impact of transposable elements in genome evolution and genetic instability and their implications in various diseases. Genomics Inform. 2014. PMID: 25317108.
The impact of transposable elements on gene functionality and genomic structural variations. Genesis. 2020. PMID: 33230956.
The impact of transposable elements on mammalian development. Development. 2016. PMID: 27875251.
The important contribution of transposable elements to phenotypic variation and evolution. Curr Opin Plant Biol. 2022. PMID: 34883307.
The Intricate Evolutionary Balance between Transposable Elements and Their Host: Who Will Kick at Goal and Convert the Next Try? Biology (Basel). 2022. PMID: 35625438.
The Mobilome of Reptiles: Evolution, Structure, and Function. Cytogenet Genome Res. 2019. PMID: 30739120.
The power of “controllers”: Transposon-mediated duplicated genes evolve towards neofunctionalization. J Genet Genomics. 2023. PMID: 37068629.
The role of transposable elements in aging and cancer. Biogerontology. 2023. PMID: 37017895.
The role of transposable elements in health and diseases of the central nervous system. J Neurosci. 2013. PMID: 24198348.
The Role of Transposable Elements of the Human Genome in Neuronal Function and Pathology. Int J Mol Sci. 2022. PMID: 35628657.
The Structural, Functional and Evolutionary Impact of Transposable Elements in Eukaryotes. Genes (Basel). 2021. PMID: 34203645.
Transposable Element Dynamics and Regulation during Zygotic Genome Activation in Mammalian Embryos and Embryonic Stem Cell Model Systems. Stem Cells Int. 2021. PMID: 34691189.
Transposable Element Mediated Innovation in Gene Regulatory Landscapes of Cells: Re-Visiting the “Gene-Battery” Model. Bioessays. 2018. PMID: 29206283.
Transposable Elements and Human Diseases: Mechanisms and Implication in the Response to Environmental Pollutants. Int J Mol Sci. 2022. PMID: 35269693.
Transposable elements and their role in aging. Ageing Res Rev. 2023. PMID: 36773759.
Transposable elements as a potent source of diverse cis-regulatory sequences in mammalian genomes. Philos Trans R Soc Lond B Biol Sci. 2020. PMID: 32075564
Transposable Elements as a Source of Novel Repetitive DNA in the Eukaryote Genome. Cells. 2022. PMID: 36359770.
Transposable elements as essential elements in the control of gene expression. Mob DNA. 2023. PMID: 37596675
Transposable elements as genetic accelerators of evolution: contribution to genome size, gene regulatory network rewiring and morphological innovation. Genes Genet Syst. 2020. PMID: 31932541.
Transposable elements as new players in neurodegenerative diseases. FEBS Lett. 2021. PMID: 34626428.
Transposable Elements Co-Option in Genome Evolution and Gene Regulation. Int J Mol Sci. 2023. PMID: 36768929.
Transposable elements in Drosophila. Mob Genet Elements. 2017. PMID: 28580197.
Transposable Elements in Pluripotent Stem Cells and Human Disease. Front Genet. 2022. PMID: 35719395.
Transposable elements shape the evolution of mammalian development. Nat Rev Genet. 2021. PMID: 34354263.
Transposable elements, genome evolution and transgenerational epigenetic variation. Curr Opin Genet Dev. 2018. PMID: 29525544.
Transposable elements: all mobile, all different, some stress responsive, some adaptive? Curr Opin Genet Dev. 2018. PMID: 29705597.
Transposable elements: genome innovation, chromosome diversity, and centromere conflict. Chromosome Res. 2018. PMID: 29332159.
Transposable Elements: Major Players in Shaping Genomic and Evolutionary Patterns. Cells. 2022. PMID: 35326499.